TrioのMRSカーブフィットソフトについて (編集途中)
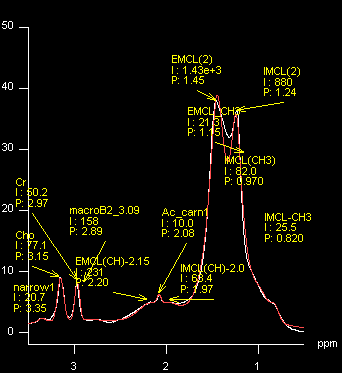
TrioのMRS処理ソフトではベースライン処理が一つのスプライン関数で行われているため、全体を一度に処理するには向きません。しかし、部分的にピークをフィットするためには十分な機能です。脳のような高分子やリン脂質などの波形が複雑に重なったスペクトルではLCModelのようなベースライン推定をして差し引くことでピークフィットが可能になるが、骨格筋のような比較的シンプルなスペクトルではTrioで行うフィティングでもかなりよい結果が得られる。運動後の骨格筋に出現するacetylcarnitineの2.13 ppmのピークのフィッティングを例に説明する。(Magn. Reson. Med. 2012のJimin Renらの7Tでの報告を参考にして欲しい。)
比較的フィットのよいスペクトルのフィットサンプル例を以下に示す。
このフィッティング結果は以下のようになります。
| Metabolite | Pos/ppm | Integral | Ratio |
| IMCL(CH3) | 0.94 | 44.71 | 0.79 |
| IMCL-CH3 | 0.83 | 15.97 | 0.28 |
| EMCL_CH3 | 1.1 | 12.77 | 0.23 |
| IMCL(2) | 1.24 | 565.49 | 10.02 |
| EMCL(2) | 1.41 | 2010.62 | 35.61 |
| IMCL(CH)-2.0 | 2 | 55.14 | 0.98 |
| Ac_carn1 | 2.07 | 1.86 | 0.03 |
| EMCL(CH)-2.15 | 2.2 | 222.66 | 3.94 |
| Cr | 2.96 | 56.46 | 1 |
| macroB2_3.09 | 2.87 | 91.12 | 1.61 |
| Cho | 3.14 | 84.28 | 1.49 |
| narrow1 | 3.33 | 52.39 | 0.93 |
| narrow3.8 | 3.84 | 9.45 | 0.17 |
| narrow3_3.9 | 3.87 | 12 | 0.21 |
| Cr2 | 3.89 | 6.69 | 0.12 |
| macroB1 | 4.42 | 363.97 | 6.45 |
| Water | 4.63 | 302.99 | 5.37 |
| glycerol (CHO-)-5.2 | 5.25 | 18.05 | 0.32 |
| Water2 | 5.31 | 78.99 | 1.4 |
| IMCL(H=3D)-5.3 | 5.4 | 89.36 | 1.58 |
| EMCL (H=3D)-5.45 | 5.49 | 33 | 0.58 |
| Carn_C4a_6.8 | 6.99 | 1.2 | 0.02 |
| Carn_C2a_7.9 | 7.95 | 1.65 | 0.03 |
このフィッティングのプロットコールはメールでお問い合わせください(umeアットマーク+大学ドメイン(webアドレスからwwwを除く))。
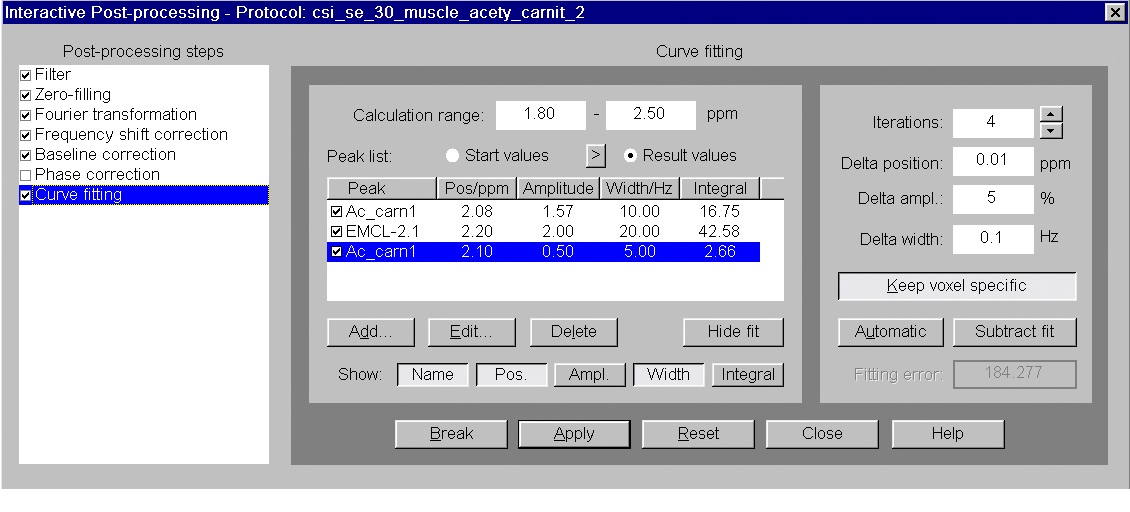
フィッティングカーブの設定方法
骨格筋を測定すると2 ppm 付近には山のような信号が認められ、ベースラインを一つのスプライン関数でフィットすることは難しい。このため、1.8 -2.7 ppmの範囲を拡大し、3次のスプライン関数でフィットする。除外領域を1.9-2.45 ppmとした。特にこの領域にはacetylcarnitineのCOO-CH3のピーク以外にも脂肪酸のCH2のピークも重なるので少し複雑になっている。この領域で主に3つのピークをフィッティングさせる必要がある。フィッティングテーブルを編集して3つの新しいピークを作成しておく。幸いにacetylcanitinはシャープなピークであり、CSIで5 mm角程度の領域とすると5-10Hz程度でフィットできる。一方脂肪酸由来のピークは幅広になる。
また、新しいピークとして(1) 3.12 ppm – 3-10 Hz、(2) 2.2 ppm- 10-30 Hz (EMCLa), (3) 2.4 ppm – 15-30 Hz (EMCLb) を作成し、データに作用させます(openする)。(必ず各フィッティング結果をチェックが必要)
settingを開く。
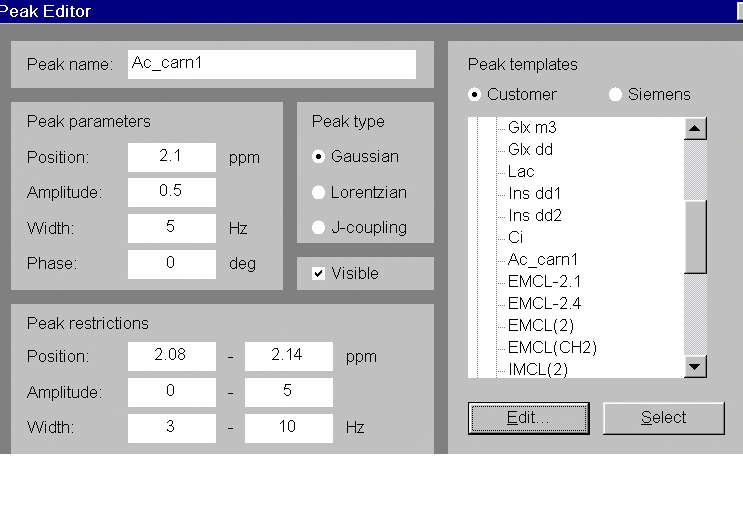
次に、peak list下のEditをクリック、
Add peakで必要な値を入力しメニューからSaveをする。
fittingの結果以下のように見ることができる。ここでフィッティングの状態をチェックする。
テップ1: chemical shiftを3.0 ppmのピークを使って合わせる
ステップ2: 領域を拡大する
ステップ3: ピークをfittingする
複数のピークを持つ代謝物質のフィッティングについて
グルコースの信号を例に説明します。
D-グルコースの水溶液の信号は図のように3ppから4ppmのピーク群と5.2-5.3ppmにピークが認められます。(クレアチンのピークを3.02ppmとしています。)
各ピークのケミカルシフトと相対強度を調べてテーブルにします。
次にケミカルシフトをHzに直して起きます。
テーブルができたら、Curbe fittingのwindowでAddをクリックします。
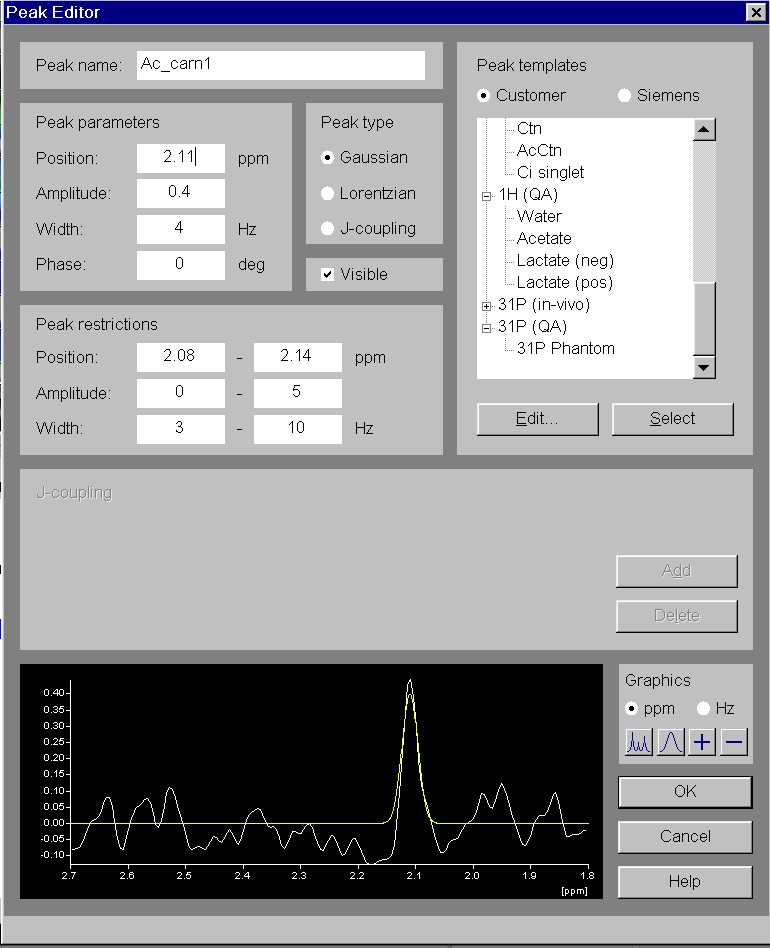
Edit Pieakをクリックします。
Add peakをクリックします。
ここではPeak folderにglucose_multiと入力しました。
J-coupingを選択し、
J-coupingのとろでAddをクリックします
Nmaにgloco_3.12と入力しました。Pos/Hzには-80.0, Amp は0.1, Width 0.3(※0-1.0を入力), Phse 0, TypeはLorentzian(※スペースを間違えるとエラーで何も表示されない)を入力します。3.12ppmのところに黄色のピークが表示されるます。
次にAddをクリックして、以下のパラメータを入力します。
gulco_3.20 -71.2 0.22 0.3 0
gluco_3.28 -60.6 0.33 0.3 0
gluco_3.32 -50.6 0.40 0.22 0
gluco_3.40 -46.6 1.16 0.22 0
gluco_3.42 -42.7 0.60 0.22 0
gluco_3.48 -38.5 0.70 0.20 0
gluco_3.51 -32.4 0.55 0.20 0
gluco_3.53 -29.0 0.74 0.23 0
gluco_3.60 -20.3 0.70 0.23 0
gluco_3.67 -10.5 0.95 0.20 0
gluco_3.80 -0.40 3.75 0.27 0
gluco_3.82 6.50 0.73 0.26 0
gluco_3.86 15.00 1.10 0.30 0
gluco_5.18 174.0 0.60 0.25 0
gluco_5.20 178.0 1.10 0.25 0
gluco_5.23 182.0 0.40 0.25 0
下のOKをクリックします。
Peak listのPeakのリストのチェックボックスのチェックを入れて右のAutomaticをクリックしてフィッティングを実行します。